Бионическую бактерию научили синтезировать белки с несколькими «ненатуральными» аминокислотами
Штамм бактерий с расширенным генетическим кодом, разработанный учеными Института биологических исследований Солка (Salk Institute for Biological Studies), может проложить путь к новым синтетическим медицинским препаратам и новым способам производства лекарств и биотоплива.
Впервые ученым удалось создать бактерию, способную эффективно встраивать «ненатуральные» аминокислоты – искусственные дополнения к 20 аминокислотам, используемым в качестве биологических строительных блоков – одновременно в несколько сайтов синтезируемых ею белковых молекул. Этот метод может стать мощным инструментом как для изучения биологических процессов, так и для создания инженерных бактерий, производящих синтетические химические вещества.
Генетически модифицированные бактерии уже используются для производства медицинских препаратов, например, синтетического инсулина. Этот способ получения инсулина, необходимого для снижения уровня сахара в крови больных диабетом, уже в значительной степени заменил собой производство инсулина из поджелудочной железы животных.
Но на сегодняшний день технологии с использованием рекомбинантной ДНК используют только природные аминокислоты, что ограничивает функции получаемых в результате белковых продуктов. Возможность встраивать в белки ненатуральные аминокислоты – Uaas (unnatural amino acids) – могла бы значительно расширить и свойства белков, и область их применения.
В 2001 году, «расширив генетический код», доцент лаборатории химической биологии и протеомики Института Солка Лэй Ван (Lei Wang) и его коллеги впервые создали бактерию Escherichia coli, способную встраивать в синтезируемые ею белки не встречающиеся в живых организмах аминокислоты, а в 2007 году ученые применили разработанный ими метод на клетках млекопитающих.
Добавление Uaas изменяет химические свойства белков, обещая новые перспективы их использования в научных исследованиях, разработке лекарств и химической промышленности.
Так, Ван и его коллеги встроили в белки Uaas, светящиеся под микроскопом при воздействии на них светом с определенной длиной волны. Так как белки служат основой широчайшего спектра клеточных функций, способность визуализировать эти молекулярные машины в живых клетках в реальном времени поможет ученым расшифровать множество биологических процессов, в том числе участвующих в развитие заболеваний и связанных с процессом старения.
Чтобы добавить инструкции по встраиванию «ненатуральной» аминокислоты в генетический код бактерий, Ван и его коллеги использовали стоп-кодоны – специальные генетические последовательности в гене белка. Стоп-кодоны «приказывают» синтезирующей белок молекулярной машине прекратить добавление аминокислот к образующейся белковой цепочке. Избирательно включив в модифицированную генетическую последовательность бактерии стоп-кодон, ученые ввели в бактерию инженерные молекулы, которые хирургически встраивают молекулу Uaa у этого стоп-кодона. Это «научило» бактерию вырабатывать белки с искусственной аминокислотой, встроенной в их основу.
Но получение таких белков с помощью стоп-кодонов ограничивается одним серьезным препятствием: одновременно в белок можно встроить только одну искусственную аминокислоту.
Проблема состоит в том, что еще один биологический «игрок» – белок, известный как фактор освобождения полипептидных цепей, или релиз-фактор 1 (release factor 1, RF1) – слишком рано останавливает синтез содержащего Uaa белка. Хотя ученые могут встроить стоп-кодоны для Uaas во многие участки генетической последовательности, RF1 все равно оборвет сборку белка у первого же стоп-кодона, предотвращая синтез длинных белков, содержащих несколько искусственных аминокислот.
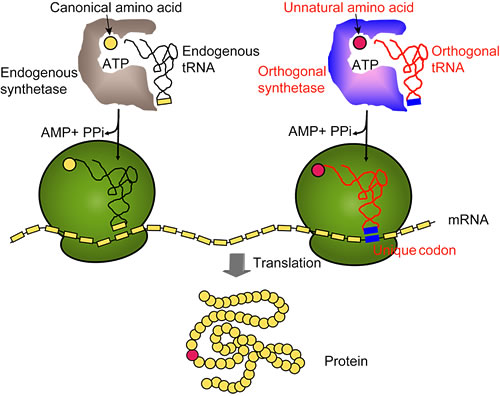 Генетическое включение Uaa в живую клетку. Инженерная ортогональная синтетаза связывает
Генетическое включение Uaa в живую клетку. Инженерная ортогональная синтетаза связывает
желательную Uaa с ортогональной тРНК, тРНК доставляет Uaa к уникальному распознаваемому ей кодону
(стоп-кодону). (Рис. wang.salk.edu)
В своей статье, опубликованной в Nature Chemical Biology, ученые описывают, как им удалось обойти это ограничение. Они удалили ген, кодирующий RF1, препятствующий синтезу длинных белков, содержащих Uaas. Но, так как E. Coli не может жить без RF1, им пришлось изменить выработку другого «игрока» – релиз-фактора 2 (RF2) – таким образом, что он мог спасти инженерную бактерию.
В результате был получен штамм бактерий, способных эффективно синтезировать белки, содержащие несколько Uaas. Эти синтетические молекулы перспективны для разработки лекарственных препаратов с намного более широкими биологическими функциями, чем те, что можно создать с использованием белков, состоящих только из природных аминокислот. Они могут служить основой и для производства промышленных растворителей, биотоплива и т. д. и помочь в решении экономических и экологических проблем, связанных с основанными на нефти промышленностью и транспортом.
«Это первый случай получения жизнеспособного штамма бактерий с такими свойствами. Это делает использование искусственных аминокислот в биологической инженерии гораздо ближе к реальности и открывает много новых возможностей – от создания лекарств с более длительным периодом разрушения в крови до производства химических веществ экологически безопасным способом», – комментирует перспективы использования разработанного им метода Лэй Ван.
Еще о работе Лэй Вана
«Ненатуральные» аминокислоты помогают ученым изучать биологию стволовых клеток и функции белков
Аннотация к статье
RF1 knockout allows ribosomal incorporation of unnatural amino acids at multiple sites
- Источник(и):
- Войдите на сайт для отправки комментариев
 Сайт о нанотехнологиях #1 в России
Сайт о нанотехнологиях #1 в России