Компьютерное моделирование помогает увидеть механизмы эволюции
Используя мощные компьютеры для моделирования белковых процессов, ученые Государственного университета Флориды (FSU) открыли новый механизм, позволяющий по-новому взглянуть на процесс эволюции на молекулярном уровне. Понимание таких процессов дает возможность создания новых и более эффективных антипаразитарных лекарств.
Вей Янг (Wei Yang), профессор факультета химии и биохимии в FSU, а также ведущий сотрудник Института молекулярной биофизики в FSU, при участии сотрудников Duke University и Brandeis University недавно создал поразительные компьютерные модели белковых молекул инозин монофосфат дегидрогеназы (IMPDH). Этот белок контролирует метаболические процессы, связанные с ДНК и РНК и влияет на способность биологической системы к быстрому самовоспроизводству.
«Создав такие модельные системы IMPDH, мы увидели нечто не известное ранее, – рассказывает Янг. – Изначально ученые предполагали, что существует единственный путь катализа метаболических процессов, однако IMPDH оказалась способна обеспечивать еще один механизм, в результате которого могут происходить подобные изменения. Второй процесс несколько менее продуктивен в клетках, но все равно происходит».
 Рис.1. Профессор факультета Химии
Рис.1. Профессор факультета Химии
и Биохимии FSU Wei Yang (слева)
и старший научный сотрудник Института
Молекулярной Физики Donghong Min
Почему же один белок управляет двумя путями одного и того же процесса? Янг и его коллеги полагают, что более медленный механизм оказался «побочный продуктом» в эволюционных изменениях работы белка.
«Это открытие можно считать значимым по нескольким причинам, – рассказывает Янг. – В первую очередь, оно предлагает задуматься об эволюционных процессах в структуре белковых молекул. Обычно мы говорим об эволюции как о процессе адаптации популяций организмов к условиям окружающей среды в течение довольно длительных промежутков времени. Наши результаты показали механизм такой адаптации на молекулярном уровне, и такое рассмотрение помогает ученым видеть процесс эволюции «изнутри».
Наши результаты представляют собой также успешную попытку компьютерного моделирования биологических процессов. Сначала мы строим процесс на компьютере, предполагая и предсказывая его особенности, а затем проверяем полученные результаты в условиях реальных лабораторных экспериментов. Те результаты, которые нам удалось увидеть, говорят о расширении возможностей ученых в ответе на вопросы о функционировании клетки на молекулярном уровне.
Поскольку IMPDH является ключевым регулятором некоторых процессов, мы можем сконцентрироваться на поиске лекарств, нацеленных на взаимодействие именно с IMPDH мишенью, такие лекарства будут иметь антипаразитарные свойства».
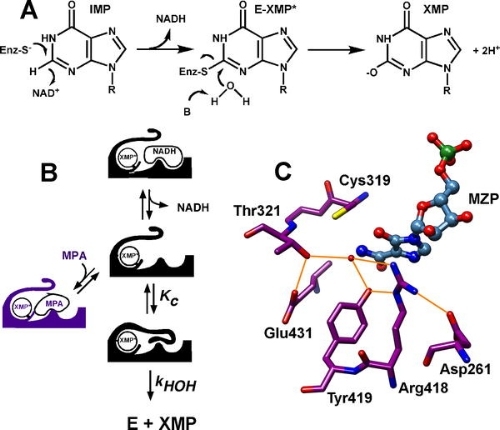 Рис.2. Каталитический механизм IMPDH. А) Биохимическая реакция; В) Конформационные превращения IMPDH; С) Структура каталитического центра белка
Рис.2. Каталитический механизм IMPDH. А) Биохимическая реакция; В) Конформационные превращения IMPDH; С) Структура каталитического центра белка
Декан факультета Химии и Биохии в FSU Joseph Schlenoff признает, что подход Янга очень удачный, поскольку он основан на четких предположениях и скурпулезных расчетах и может описывать сложнейшие процессы в реальных биологических системах. Безусловно, это открывает большое будущее в использовании учеными компьютерного моделирования.
Команда ученых, проводивших данное исследование, этим летом опубликовала свои открытия в работе An Enzymatic Atavist Revealed in Dual Pathways for Water Activation в журнале cо свободным доступом PLoS Biology.
Профессор Стэнфордского университета Dan Herschlag, редактировавший эту публикацию, также воодушевлен ее новаторским исследовательским качеством: «Работа проясняет основные аспекты нашего понимания того, как белок работает и как он произошел. Эта работа подготовила мощное воссоединение компьютерного и экспериментального методов и является примером междисциплинарного подхода к ответам на важные вопросы».
Мария Костюкова
- Источник(и):
-
Computational Biochemist Uncovers Molecular Clue to Evolution
-
An Enzymatic Atavist Revealed in Dual Pathways for Water Activation
- Войдите на сайт для отправки комментариев
 Сайт о нанотехнологиях #1 в России
Сайт о нанотехнологиях #1 в России